Deep learning: intelligenza artificiale al servizio della biologia
Ph: naadispeaks.wordpress.com
L’idea dei ricercatori
Tutto ha avuto inizio circa quattro anni fa, quando i ricercatori di Google Accelerated Science, una divisione di ricerca di Mountain View, in California, che mira a usare le tecnologie di Google per accelerare le scoperte scientifiche, si sono rivolti al gruppo di Steve Finkbeiner al Gladstone Institute of Neurological Disease a San Francisco. La loro idea era quella di applicare “l’apprendimento profondo” (deep learning) all’enorme numero di immagini generate dal laboratorio di neuroimaging in questione.
Il gruppo di Finkbeiner infatti produce valanghe di dati grazie a una strategia di imaging ad alta velocità nota come microscopia robotizzata, sviluppata per lo studio delle cellule cerebrali. Il gruppo però non era in grado di analizzare i dati alla stessa velocità con cui li acquisiva; così Finkbeiner ha accolto con favore l’opportunità di collaborare con Google.
Il gruppo di Finkbeiner e gli scienziati di Google hanno creato un algoritmo di apprendimento profondo usando due serie di cellule, una marcata artificialmente per evidenziare le caratteristiche che di norma gli scienziati non riescono a vedere, l’altra non marcata. Quando in seguito l’algoritmo ha analizzato immagini di cellule non marcate che non aveva mai visto prima, dice Finkbeiner,”è stato sorprendentemente bravo a prevedere quali avrebbero dovuto essere le marcature per quelle immagini”.
Deep learning – come apprendono le macchine
Gli algoritmi di deep learning si basano su reti neurali, un modello computazionale proposto per la prima volta negli anni Quaranta, in cui strati di nodi neuronali imitano il modo in cui il cervello umano analizza le informazioni.
Gli algoritmi acquisiscono quindi informazioni da un insieme di dati estremamente ampio e classificato (come una raccolta di immagini o genomi), e le usano per creare uno strumento predittivo basato su schemi sepolti nello stesso insieme di dati. Una volta allenati, gli algoritmi possono usare questo addestramento per analizzare altri dati, a volte provenienti da fonti differenti. Il modello così creato sarà capace di riconoscere rapidamente ed in modo sempre più preciso e discriminante i dati forniti.
Deep learning – l’applicazione in biologia
La classificazione di immagini e l’analisi di grandi sequenziamenti genomici, sono sicuramente i due maggiori target che si vuole raggiungere attraverso l’uso degli algoritmi di deep learning.
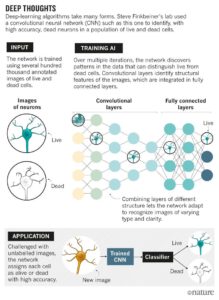
Nel caso della predizione di immagini si usano le convolutionl neural network. L’algoritmo suddivide ogni immagine in porzioni topologicamente compatte ciascuna della quali sarà processata da filtri in modo da ricercare pattern specifici.
Questo procedimento produce un insieme di feature maps (mappe di attivazione) per i vari filtri. Sovrapponendo le varie feature maps di una stessa porzione di immagine otteniamo un volume di output. Cioè un valore di probabilità per ogni neurone in output.
Questo sarà lo schema generale a cui verrà sottoposta ogni immagine in input e ad ogni nuovo dataset ci sarà un ricalcolo delle probabilità finali in modo da ottenere sempre più un risultato attendibile.
Nature.com
Riguardo ad i dati di sequenziamento si possono avere numerosi approcci derivati dai diversi input che possono venire dati. Utilizzare DNA o RNA è chiaramente una prima grossa differenza che necessita di approcci sensibilmente diversi, così anche come strutture complesse ottenute da DNA-proteina o DNA-RNA.
Fra i tanti algoritmi è stato sviluppato anche DeepVariant (GitHub) da Verily Life Sciences a San Francisco. Il programma traduce le informazioni genomiche in rappresentazioni simili a immagini, che poi sono analizzate proprio come immagini. In questo modo si cerca di utilizzare metodi più consolidati per l’analisi piuttosto che realizzare strategie ex novo. Grazie a DeepVariant si sono raggiunti tassi di errore del 2% invece del 20 % tipico di altri approcci.
Cosa accadrà in futuro – rischi e innovazione
Come con qualsiasi tecnica di biologia computazionale, i risultati che derivano dagli algoritmi sono “buoni” solo quanto lo sono i dati in ingresso. Gli algoritmi di apprendimento profondo richiedono insiemi di dati estremamente grandi e ben classificati, in modo che gli algoritmi possano imparare a distinguere le caratteristiche e categorizzare i modelli. Finkbeiner nota che nel suo lavoro la messa a punto di un algoritmo migliora significativamente dopo circa 15.000 esempi.
Per aggirare la sfida, i ricercatori hanno iniziato a lavorare su come ottenere di più con meno dati. Gli scienziati possono anche sfruttare il transfer learning, cioè la capacità delle reti neurali di applicare a un certo tipo di dati alcune abilità di classificazione acquisite da un tipo di dati differente.
Un’altra sfida per l’apprendimento profondo è che i computer sono indolenti e scarsamente intelligenti, nota Michelle Dimon, ricercatrice di Google Accelerated Science: non sanno distinguere differenze biologicamente rilevanti da variazioni normali.
Tuttavia, per il futuro della ricerca in campo genomico e della medicina di precisione l’impiego di strumenti computazionali sempre più potenti è fondamentale. Gli strumenti di apprendimento profondo potrebbero anche aiutare i ricercatori a stratificare i tipi di malattie, capire le sottopopolazioni patologiche, trovare trattamenti e abbinarli ai pazienti in base ai test clinici e alla terapia.
